연구의 핵심 목표는 크론병(Crohn’s disease) 환자의 미생물 군집에서 미생물 간 상호작용과 미생물-대사 경로 관계를 그래프 모델로 분석하는 고급 생물정보학 파이프라인을 구축
용어정리..
- KEGG란
- 생명체의 유전자와 대사 기능을 연결해주는 생물정보학 디비
- 교토 유전자 및 유전체 백과사전
- 이 task 에서 KEGG 의 역할은
- 16s 데이터로 미생물 종류를 파악하고
- PICURSt 로 KO를 예측하고
- KEGG로 KO → 기능, 경로로 연결
- 미생물이 어떤 기능을 수행할 수 있는 가를 알고 싶은거니까
- OTU
- 운영 분류 단위?
- 미생물 시퀀싱 데이터를 기반으로, 비슷한 유전자 서열끼리 묶은 그룹을 의미
- 이 개념이 필요한 이유는?
- 현실적으로 모든 미생물의 정확한 종을 파악하기는 어려우니까
- 뭐 유전자 서열이 97% 이상 비슷하면 같은 종으로 간주하자~ 라는 의미
- LEfSe
- 분석단순히 통계적 유의성 뿐만 아니라 “ 그 차이가 얼마나 강한가”(effect size) 도 같이 보여준다는거임
- 비모수적 크루스칼-왈리스 검정?
- 그룹 간에 차이가 있는 특징을 찾는 과정임
- LDA
- 그룹 간 차이의 영향력을 정량화 하는 과정
- 비모수적 크루스칼-왈리스 검정?
- 두가지 통계적 단계로 구성됨
- 다양한 그룹 사이에서 유의미하게 다른 미생물 또는 기능적 특징을 찾는 도구임
Background
- 마이크로바이옴은 인간의 건강에 중요한 역할을 하며, 특히 장내 미생물은 면역조절, 대사, 점막 항상성 유지에 관여한다
- 크론병은 이러한 장내 미생물군의 불균형과 관련이 있지만, 구체적인 메커니즘은 명확하지 않음
- 기존 연구는 통계 상관분석에 의존
- 미생물 상호작용을 정량적으로 모델링하기 어려움
- 따라서 복잡한 미생물 생태계 관계를 효율적으로 표현할 수 있는 새로운 그래프 모델이 필요
- 생물학적 접근은 단순히 미생물 간의 상호작용뿐만 아니라, 이러한 상호작용이 숙주 생물의 건강과 기능에 어떻게 영향을 미치는지까지 포괄적으로 모델링하고자 함
Method
두 종류의 관계(내부 상호작용과 외부 표현형 관계)를 각각 다른 엣지 그룹으로 구분해 표현할 수 있는 특수한 그래프 구조
- 미생물 풍부도 데이터와 해당 미생물의 유전체 정보를 결합해서
- 미생물간 내부 상호작용
- 미생물과 대사 경로 간 외부 연관성
을 나타내는 독립적인 네트워크를 생성
2. 위에서 생성된 넨트워크로 스플릿 그래프를 구성함
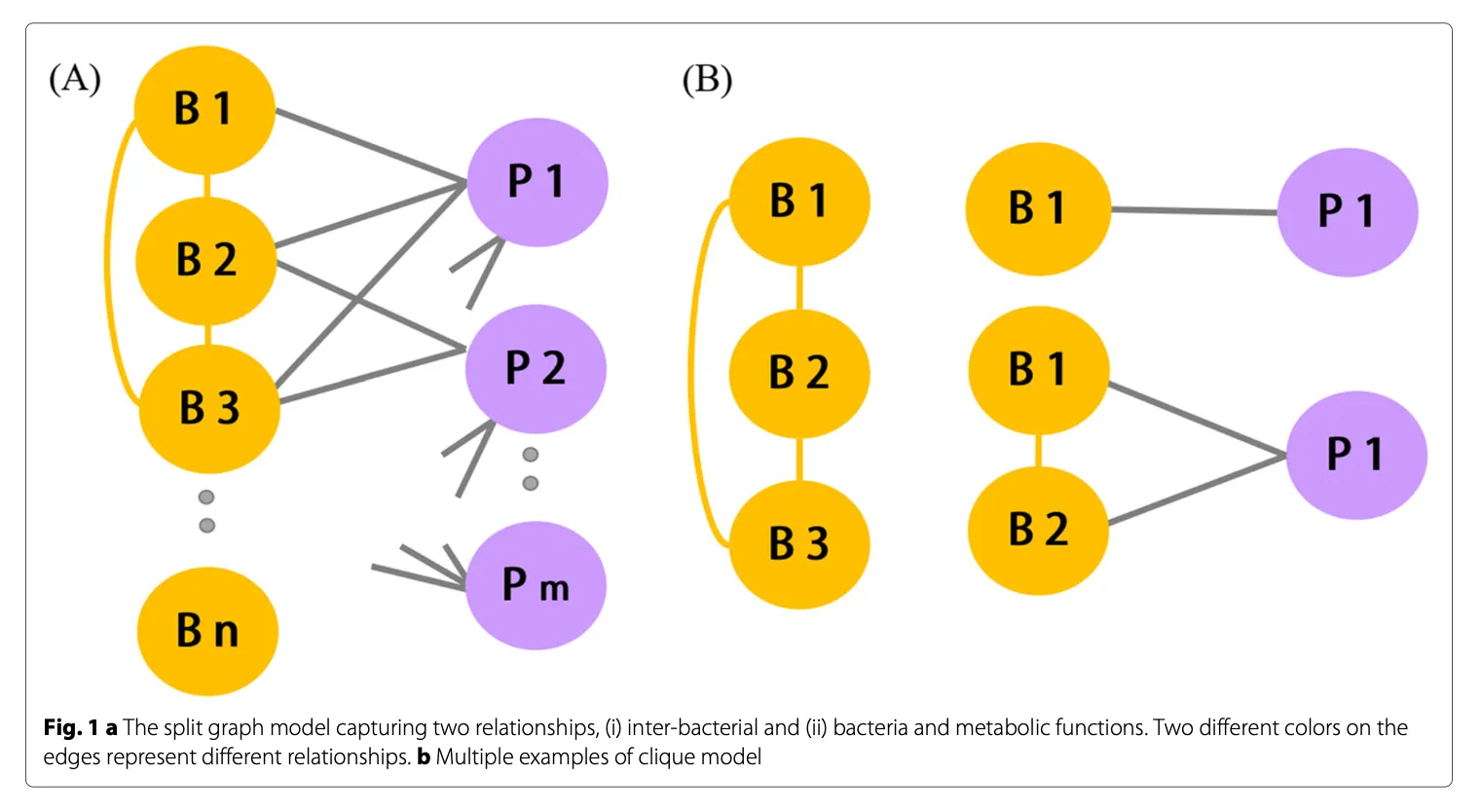
Split graph model
- 스플릿 그래프는 노드를 두 개의 집합으로
- 클리크 Q : 서로 강하게 상호작용하는 미생물
- 모든 노드가 서로 연결되어 있는 완전 부분 그래프
- 독립집합 I : 미생물의 기능적 표현 → 대사 경로를 의미
- 서로 간에 연결된 엣지가 없는 노드 집합
- 클리크 Q : 서로 강하게 상호작용하는 미생물
- 두 개의 엣지 집합
- 클리크 엣지 : Q 내 노드들 간의 엣지
- 미생물 간의 상호작용
- 크로스 엣지 : Q 의 노드와 I 의 노드를 연결하는 엣지
- 미생물과 대사경로 간의 관계
- 클리크 엣지 : Q 내 노드들 간의 엣지
- 클리크 간의 엣지는 미생물 간의 강한 상관관계를 의미
- 클리크-독립집합 간의 엣지는 미생물과 대사경로의 연관을 의미
- 각 엣지에는 상관계수 또는 KO (KEGG Orthologs) 기반의 밀도 값을 가중치로 부여
- 노란색 원
- 미생물 노드
- 보라색 원
- 대사 경로 또는 표현형 노드
- 클리크 내 노드간 엣지
- 미생물 간 강한 상관관계
- 크로스 엣지
- 미생물 ↔ 대사 경로 엣지
→ 엣지에는 가중치를 부여하여, 단순한 연결 이상으로 co-occurence나 상관관계 강도를 표현할 수 있음
- 독립 노드(대사경로)는 서로 연결되어있찌 않음 !!!! !
- 실제 연관성이 있더라도 표현하지않음
- 이유는?
- 이 기능에 영향을 주는 미생물은 누구인가????? 만 알고 싶기 때문
- 기능끼리 연결된 정보는 지금 질문에 중요하지 않음
⇒ 결과적으로 가중치가 높은 최대 클리크는 다음을 의미함
- 하나의 대사 경로와
- 해당 경로에 영향을 주는 서로 강하게 연결된 미생물 집단
→ 핵심 바이오마커 모듈에 해당
Data processing of 16s rRNA gene sequence datasets
- 16s rRNA
- 크론병 환자의 점막 조직 샘플 36 개 (CDT)
- 크론병 환자의 대변 샘플 10 개 (CDS)
- 건강한 개인의 대변 샘플 9 개 (HCS)
- QIIME 파이프라인 사용
- 97 퍼센트 유사도 기준으로 클러스터링
- Greengenes DB를 사용해서 미생물 classification을 수행
Metagenome prediction and metabolic reconstruction of 16S rRNA datasets
- PICRUSt v 1.1.0 을 사용해서 메타 유전체를 예측
- 16s rRNA 계통수로부터 OTU를 해당 유전자 내용물에 연결할 수 있도록 도와줌
- 이 예측 과정이 필요한 이유
- 16s rRNA 시퀀싱은 미생물의 종류는 알려주지만, 그 미생물이 어떤 유전자를 갖고 있는지는 직접 알려주지않음
- 하지만 미생물이 수행하는 기능(대사경로)를 알고싶으니까 유전자 정보를 간접적으로 예측해야해
- 그래서 PICRUSt를 사용
- 관찰되지않은 유전자 상태를 계통수 기반으로 재구성한다 라느 ㄴ의미
- 그래서 PICRUSt를 사용
- PISRUSt 내장 알고리즘으로 16s rRNA 계통수로부터 OTU를 해당 유전자 내용물에 연결할 수 있도록 도와줌
- 이후, 예측된 KO 기능 프로파일을 기반으로 HuMAnN2 파이프라인을 활용하여 KEGG 대사경로를 재구성
- KO는 각 유전자의 역할, KEGG 대사경로는 그 유전자들이 속한 생화학적 경로를 의미
Detection of taxonomic and metagenomics biomarkers
- LEfSe 도구를 사용해서
- 3 가지 그룹(CDT, CDS, HCS) 에서 가장 생물학적으로 정보량이 많은 특징들을 식별
- 분류학적 조성이나 기능적 대사경로 등의 특징들!
- 3 가지 그룹(CDT, CDS, HCS) 에서 가장 생물학적으로 정보량이 많은 특징들을 식별
Network construction and split graph analysis
- 미생물 간 상호작용 탐지특정 조건을 만족하는 공출관계만 선택
- CDT, CDS, HCS 내에서 미생물 간의 공출 패턴을 드러내는 미생물 간의 상관관계를 분석
- 미생물과 대사 경로 간의 상관관계 탐지
- 공출관계가 있는 미생물들에 대해 그 풍부도와 KO 사이의 상관관계를 분석
- 상관계수 rho > 0.6
- FDR 보정 p-value < 0.05
- 특정 미생물과 KEGG 대사 경로 사이의 연관성을 추정
- 해당 미생물과의 연관된 KO 들의 수
- 해당 경로에 포함된 전체 KO의 수로 나눈 비율로 계산
- 공출관계가 있는 미생물들에 대해 그 풍부도와 KO 사이의 상관관계를 분석
- 두 단계로 이루어짐
- 네트워크 구성 및 스플릿 그래프 생성
- 미생물 간 상호작용 → 클리크 엣지
- 서로 강하게 연결된 미생물 집단이 클리크
- 미생물 ↔ 대사 경로 관계 → 크로스엣지
- 각 미생물이 어떤 대사경로에 관여할 가능성이 높은지를 정량적으로 표현
- 두가지 관계를 통합함
| 작성자 | 김윤희 |
| 소 감 | 오늘은 읽어볼 논문을 리뷰하는 시간을 가졌다. 정해진 모각코 시간에 읽으려고 하니 미뤄지지 않고 잘 읽을 수 있어서 좋았다, |
| 일 시 | 2025. 5. 9. (금) 18:00 ~ 21:00 |
| 장 소 | 미래관 429호 자율주행스튜디오 |
| 참가자 명단 | 신수민, 임혜진, 배세은, 김윤희 (총 4명) |
| 사 진 |  |
'윤희' 카테고리의 다른 글
| [호붕싸 모각코 9차] Show and Tell code 구현 (0) | 2025.05.02 |
|---|---|
| [호붕싸 모각코 8차] 컴퓨터네트워크 복습 (0) | 2025.04.11 |
| [호붕싸 모각코 7차] High-Resolution Image Synthesis with Latent Diffusion Models 논문리뷰 (0) | 2025.04.04 |
| [호붕싸 모각코 6차] CA-MoE: Channel-Adapted MoE for Incremental Weather Forecasting 논문 리뷰 (0) | 2025.03.31 |
| [호붕싸 모각코 5차] beakjoon 2225. 합분해 (0) | 2025.03.24 |